こんな感じの
ついでに、講師のgitHubから飛んで買えば安くなるかも?よくわかんないけど
というわけで、環境構築
動画だと、Anaconda使うみたいっすね
ただ、動画通りにやっても面白くないし、自分のパソコンにはすでにAnaconda入ってるし
そして何より、環境分離したいし
てことで、今回はDockerつかってやろうかなーと
(現在2018/10/13)
(基本すべての作業をPowerShellで行っています)
今回の最終目標
1.Python3.7が使えるlinux環境を作る
2.Anacondaを使えるようにする
3.Jupyter notebookを使えるようにする
4.docker内でやる
まずは、Python3.7が使えるimage作り
DockerHubから公式のPython3.7の環境持って来ました
dockerのimageだけダウンロードするならpowershellとかで
docker pull python
でいいんですけど、せっかくだから、Dockerfile使おうかと
<Dockerfileとdocker-compose.ymlについて>
Dockerfile → imageを作るときの設定ファイル
docker-compose.yml → コンテナを作るときの設定ファイル
まずは、どんなimageを作るか考える
・とりあえずpython3.7をインストール
・pytest欲しいなぁ・・・image作るときにインストールしとこ
・作業用ディレクトリー欲しい
・・・意外と書くことなかった

これでおっけー
今回はコンテナ内で作業するつもりなので、CMDとかは使わないです
あと、ファイルはマウントホスト環境からマウントするつもりですので、COPYとかも使わないです
docker image build -t [作りたいimageの名前]:latest [Dockerfileのあるディレクトリー]
Dockerfileができたら上のコマンドでビルドします
自分の場合は、
docker image build -t ykdr/pyboot:latest .
ビルドができたら、コマンド
docker images
でちゃんとビルドできているか確認してくだせえ
さて、dockerのimageはできたので、どうやって起動するか考えましょう
基本的な方針は、WindowsのホストOSの方に、ファイルは置いておいて、実行環境だけDockerに移すつもりでいます(Windowsでセットアップした使いなれているエディタを使いたいので)
とりあえずデスクトップに作業ディレクトリーを作りまして
そこに、complete-python-bootcampの講師のgithubのレポジトリーをクローンします
Complete-Python-3-Bootcampというファイルができるので、このファイルをDocker内のPython実行環境からアクセスできるようにします
docker run --name [コンテナの名前] -i -t [イメージの名前] -v [ホスト側の作業ディレクトリーのパス]:[コンテナ内の作業ディレクトリーのパス] /bin/bash
と、コマンドで起動することもできますが、毎回マウントするパスを書いたりするのめんどくさいです
というか、コマンド長すぎて私には覚えられないです
というわけで、Docker-compose.ymlファイルを使って起動条件をまとめておきます

こんな感じで
container_name:起動するコンテナの名前
image:使うイメージの名前
tty:起動したあとすぐに終了しないためのおまじない
volumes:[ホスト側の作業ディレクトリー]:[コンテナ内の作業ディレクトリー]
ついでに、今の作業ディレクトリーの様子
準備完了!
docker-compose up
起動!

こんな感じで立ち上がります
docker exec -it pyboot-container bash
立ち上がったコンテナにbashで入るコマンドです
ここでpythonと打てば対話形式でPythonを利用できます

Python実行環境できた。
ついでにpytestもインストールされてる
さてさて、次はAnacondaですね。
実は、ここまで環境構築して、udemyの講義、Anaconda使って進めることを知ったり
python3.7のdockerのimageのベースはlinux環境なので、linux版のAnacondaをインストールすれば行けるのでは?
ってことで、このimageで作ったコンテナにAnacondaをインストールしていきます

とりあえず、マウントしているディレクトリーに共有するためのファイルで「mydesk」を作りました

ちゃんとマウントされている
Anacondaのインストーラーをこのmydeskを通して、linux環境に送り、linux環境にAnacondaをインストールします
Anaconda公式ダウンロードページからインストーラーをダウンロード
・・・あれ?64bit版?32bit版?どっち?
uname -a
unixコマンドで確認。なるほど64bit版か。

mydeskにダウンロードしたインストーラーを置いて、
Anacondaの公式ページのインストール方法見ながらインストール
(bash [インストーラー] でできるけど、詳しくは割愛)
とりあえずcondaが入っていれば大丈夫なはずってことで
conda -V
でcondaのバージョン確認
おっけー大丈夫そう
docker commitでAnacondaがインストールされたコンテナをイメージにします
docker stop [起動しているコンテナ]
一度コンテナを停止
docker commit [コンテナの名前] [生成するイメージの名前]
コンテナからイメージを作るcommitコマンドを使ってimageを作ります
これでAnacondaがインストールされたlinux環境の完成ですね
さてさて、最後にjupyter notebookを使えるようにですね
Windows環境でjupyter notebookで起動すると"なぜか"ブラウザーが開いて、localhost:8888に接続され、jupyter notebookが起動します(通信系弱いのであんまりわかってないです)
同じパソコンならjupyter notebookがlocalhost:8888から接続できるんでしょう、たぶん
しかし、linux環境にはブラウザ入ってないし、windows環境から接続するにもどこに接続すればいいかわからないし
というわけで、少しづつ解決します
まず初めに、ホスト側のwindowsからコンテナに接続できるようにします
相変わらずdocker runコマンド起動時にオプションで解決できますが、めんどくさいのでdocker-compose.ymlを変更していきます

追加部分は
ports:
- 9000:8888
この部分ですね
これはコンテナ内のport8888をホストのport9000につなぎますよーってコマンドです
[ホストのポート]:[コンテナのポート]なところに注意
これでjupyter notebookをコンテナ内で起動して、8888に接続すれば完璧ですね
まあ一筋縄ではいかないんですけどね
さてさて、google先生に何かヒントがないか尋ねてみます
dockerHubの方になんか良さそうな情報発見
というのも、Anacondaの公式DockerImageあるんで、そちらのドキュメントを読んでみます(最初からこれを使えばよかったのでは...?)
Docker Hub-continuumio / anaconda3
また、Jupyter Notebookサーバーを起動して、ブラウザからAnacondaと対話することもできます(Docker Hub anaconda3 ドキュメントより抜粋)
docker run -i -t -p 8888:8888 continuumio/anaconda3 /bin/bash -c "/opt/conda/bin/conda install jupyter -y --quiet && mkdir /opt/notebooks && /opt/conda/bin/jupyter notebook --notebook-dir=/opt/notebooks --ip='*' --port=8888 --no-browser"あ、多分これだ
/opt/conda/bin/jupyter notebook --notebook-dir=/opt/notebooks --ip='*' --port=8888 --no-browser
特にこの部分ですね
おそらくこれがjupyter notebookのdocker内で起動するいい感じの方法なのでしょう
パスは通っているはずなので/opt/conda...の部分はいらないでしょう
--notebook-dirは作業ディレクトリーでしょうか
ここだけ少し変えて動かしてみます

ちょっと内容が変わった
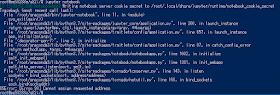
rootで開くなって言われてる?
Use --allow-rootをオプションで入れればいいのかな?

正解っぽい?
port:8888からtokenを使って接続してねって言われています

開いた!
ちゃんとdocker内のlinux環境に接続できてますねあとは、jupyter起動コマンドが長くて覚えられない問題を解決すれば完璧ですねコンテナ生成時に、jupyterを立ち上げる処理をデフォルトで入れます方針としては、docker-compose up で起動するとjupyter notebookもデフォルトで起動↓docker exec -it pyboot-compose bash で、起動しているlinux環境に接続と、こんな感じになるのが目標起動時の動作は、イメージのビルドの時に決めることができますつまり、Dockerfileをつかって、新しいイメージに作り直せばいいですね



0 件のコメント:
コメントを投稿